|
Johnson-A, Jorge-H Ing. y Est.Bio EAFIT  Resumen Los grandes volúmenes de información presentes en repositorios alrededor del planeta son el resultado de un nuevo estándar de comunicación global. Pero debido a su intenso uso, vienen presentando problemas de capacidad, y durabilidad debido a los materiales. También hay reducciones apreciables en la velocidad del acceso ocasionada por la simultaneidad de muchos usuarios. Nuevas técnicas de almacenamiento se proponen, como es el caso del uso de ADN, molécula presente en células de seres vivos con habilidades de compresión y alta durabilidad en el tiempo, y que posiblemente no tarden mucho en integrarse a los medios actuales de almacenamiento. Palabras clave Información, datos, codificación, latencia, representación, densidad, estabilidad, almacenamiento, ADN. Introducción Una de las propiedades más dominantes del cerebro de muchos animales incluida la especie Homo sapiens (humanos actuales), es su capacidad para recordar eventos y hechos del pasado. Esta característica que conocemos como recuerdos, permiten acceder a información almacenada en el pasado e incrementa la probabilidad de supervivencia dentro de un entorno concreto. Muy a pesar de la resiliencia y la velocidad de acceso a la información en el cerebro, el diseño de este órgano no nos permite hacer un almacenamiento de forma permanente, exacta y confiable. Tampoco permite que sea accedida por otras personas que la demanden, sino a través de la comunicación. Como una solución progresiva al problema de la repetición por comunicación, y a la necesidad de persistencia de la información en el tiempo, aparecen poco a poco en la historia del ser humano los registros de información, representados sobre diferentes medios y lenguajes, desde el tallado de materiales y la pintura rupestre, hasta la escritura, que aparece hace algunos miles de años. Hoy contamos con libros digitales, y datos de toda índole que son almacenamos en grandes repositorios de datos digitales, y que son denominados bajo la sombrilla del concepto de bases de datos. Pero el advenimiento de un mundo virtual hecho posible por la computación y la ubicuidad de la red internet que hoy globaliza el planeta, la presencia de una comunicación masiva de los distintos medios digitales y no digitales, junto las redes sociales, ha generado un volumen de datos de una magnitud inimaginable que desborda de forma exponencial las capacidades actuales de almacenamiento, y exige, además, más confiabilidad, más agilidad, y mayor perdurabilidad. Pero mientras el hombre intenta hallar la forma más efectiva para registrar información y así soliviar los problemas actuales de velocidad de acceso y confiabilidad de sus gigantescos repositorios digitales, se envidia cómo la naturaleza hace miles de millones de años a lo hizo. Se estima que la vida en el planeta empezó su debut hace más de 4 mil millones de años según se analiza en Dodd et al., (2017) y se sabe que requiere de la existencia de moléculas especiales para almacenar información sobre replicación y procesos vitales. Desde 1953, conocemos por los estudios de Watson & Crick (1953) sobre la estructura de la molécula de la vida, el ADN, y sabemos que con un muy corto alfabeto de 4 letras escribe frases y párrafos que describen en detalle cómo se replica, construye y opera un ser vivo. Sabemos hoy que una sola célula humana almacena en su núcleo de forma eficiente y compacta 3 mil millones de pares de bases de nucleótidos de la molécula ADN, lo que pudo ser determinado con precisión y de forma histórica en el año 2000, luego de obtenerse la secuencia detallada del genoma (International Human Genome Sequencing Consortium, 2001). Como idea de medio de almacenamiento, el genoma es posiblemente uno de los mejores medios que se conocen, pues presenta grandes ventajas en durabilidad, capacidad, y permanencia en el tiempo; entonces, se ve promisorio intentar usar el ADN como medio para almacenar otros tipos de información, y ver si es posible cubrir las deficiencias que hoy afectan el almacenamiento de nuestros datos. La naturaleza se convierte entonces en un ejemplo de cómo almacenar información de forma efectiva, durable, de bajo consumo energético, y con una capacidad envidiable. ¿Es posible entonces emular el mismo concepto para nuestros propósitos de almacenamiento de grandes volúmenes de información? Actualmente muchos investigadores se hacen la misma pregunta, y se han llevado a cabo muchos estudios tendientes a establecer la viabilidad del almacenamiento usando moléculas de ADN: si se logra hacer lo mismo que la naturaleza hace, podríamos tener una alternativa tecnológica interesante para el registro de la información. Es incierto cómo se develará el futuro del almacenamiento de la información para aliviar los problemas actuales de capacidad, velocidad de acceso y durabilidad. Pero algunas investigaciones ya orbitan alrededor de nuevos conceptos de información digital usando como medio de almacenamiento moléculas de ADN. En lo que sigue, se presentan algunas ideas de cómo es posible almacenar todo tipo de información en una molécula inicialmente destinada a indicar cómo se hace y cómo funciona un ser vivo. El almacenamiento de datos debe evolucionar Desde que el hombre comenzó a usar el arte de la pintura rupestre para recordar acontecimientos importantes para su subsistencia, la información almacenada comenzó una travesía que ha durado varios milenios a través de distintas civilizaciones y lenguas. Con la aparición de las primeras formas de escritura las cosas empezaron a mejorar en términos de registro de información: su uso en roca para guardar datos comerciales, legales y religiosos fue de gran ayuda. Pero fue la posterior aparición del papiro y el más adelante el advenimiento del papel lo que revolucionó los registros, al permitir almacenar grandes volúmenes de información de forma sencilla y condensada en rollos y folletos. Todo esto ha traído consigo una característica que se refleja subyacente a la escritura: el espacio requerido para almacenar la información que se escribe empezó a crecer hace cientos de años, lo que se refleja hoy en anaqueles en bibliotecas personales, públicas y privadas. La escritura en papel exige gran cantidad de espacio. Pero el papel no es un material inmutable y se degrada naturalmente debido a procesos bioquímicos asociados al lugar en el que se almacenan, y también a su interacción con el medio ambiente al que se expone durante su uso en la lectura. Los niveles de degradación son tales, que libros antiguos que no cuentan con copia, deben hoy ser manipulados como si se tratara de una cirugía delicada. Incluso, si hubiera copias, dichas réplicas tendrían problemas similares. Afortunadamente con el tiempo, no sólo se ha mejorado la forma en que registramos información escrita, sino que también han aparecido nuevas formas de representación de la información distintas a la escrita: la grabación de audio, los registros fotográficos, y el cine: todos muestra de cómo la información puede variar tecnológicamente su representación y almacenaje. Pero todos estos medios, al igual que la escritura de papiros y libros antiguos, requieren de espacio físico. El audio, por ejemplo, se llevaba a pulsos electromagnéticos, que luego eran representados como alteraciones de material ferromagnético sobre cintas, donde las cintas empezaron a espacio importante. La fotografía, de su lado, usaba papel especial químico con partículas de plata para el respectivo registro visual, y este papel también usaría históricamente espacio. A todos los medios de almacenamiento mencionados se les conoce como medios análogos de almacenamiento. Todos los medios, de un modo u otro, finalmente requerirán de un espacio físico que no es para nada despreciable, y que tampoco es tolerante al medio ambiente y al uso, donde constantemente se ven sometidos a deterioro por contacto con el aire, o por procesos de degradación por bacterias que gustan de estos medios para su alimentación, o simplemente por la interacción con otros materiales al momento del uso. El advenimiento de los computadores a mediados del siglo XX nos dejó en una nueva era, la digital, que trajo consigo una nueva forma de representación de la información, el denominado sistema binario (que se basa en el uso de ceros y unos -0 y 1- para representar información). Hoy, casi toda la información se almacena en formato binario en discos magnéticos, discos ópticos como el CD, el DVD, el Blue-ray y memorias de estado sólido como las tarjetas SD y los medios SSD, entre otros. En el sistema binario se pueden representar textos, sonidos e imágenes, usando sistemas de codificación específicos. Los libros, fotos, audio y videos pueden ser hoy vistos y reproducidos muchísimas veces en medios electrónicos digitales. Adicionalmente, y para aliviar los problemas de capacidad, se usan algoritmos especializados donde la información puede ser comprimida muchas veces para ocupar un espacio mucho menor. A pesar del gran logro hasta nuestros días en el almacenamiento y compresión de datos e información, la vida útil de los dispositivos de almacenamiento físico oscila, en general, entre los 5 y 25 años (Stefano, Wang y Kream, 2018, p.1), debido a que ella está ligada al deterioro de los materiales. Pero también, el gran volumen de datos al que nos estamos enfrentando cada día, hace que se requiera poder accederlos muy rápidamente debido a la gran demanda de muchos usuarios que de forma simultanea la necesitan. Es claro que el crecimiento de la información impacta directamente su almacenamiento, como mencionan Stefano, Wang y Kream (2018): “by 2040, if everything were stored for instant access flash memory chips used in memory sticks, the archive would consume 10–100 times the expected supply of microchip-grade silicon” (Stefano, et al., 2018, pag.2), y esto hace que se requieran nuevos medios y métodos de almacenamiento de información que garanticen no sólo mayor capacidad y velocidad, sino también mayor estabilidad y durabilidad en el tiempo frente al deterioro físico. La alternativa biológica Frente a la problemática de almacenamiento en cuanto a capacidad y durabilidad, una curiosa alternativa como medio de información que está siendo experimentada en la actualidad, y es el uso de ADN (Ácido desoxirribonucleico). El ADN es una molécula orgánica que se encuentra disponible en las células de los seres vivos, y cuya función es precisamente almacenar información sobre la forma de hacer y desarrollar el mismo ser vivo que la contiene. El ADN es una entidad que representa información orgánica, y por lo tanto es un medio de almacenamiento que podría servir para almacenar otros tipos de información. Además, es una molécula donde una única de sus unidades mínimas de información, denominada nucleótido, mide tan solo 0.33 nanómetros (Mandelkern, Elias, Eden & Crothers, 1981). En los sistemas de almacenamiento modernos, como comparación, un solo bit de información requiere, no sólo varios transistores, sino también un tamaño por transistor de unos 10 a 20 nanómetros, lo que hace del tamaño del bit una unidad de entre 100 y 200 nanómetros, unas 300 a 600 veces el tamaño de un nucleótido biológico. Las ventajas son interesantes de llegarse a desarrollar y generalizar métodos para almacenar información usando como medio esta molécula. Goela y Bolot (2016) lo exponen así:
La alta densidad de almacenamiento de terabytes por cada gramo de ADN, o incluso capacidades muy superiores donde la molécula “DNA can encode two bits per nucleotide (nt) or 455 exabytes per gram of single-stranded DNA” (Church, Gao, y Kosuri, 2012), plantea una salida sorprendente al tema de almacenamiento. Comparemos estos datos de almacenamiento por gramo de peso del genoma, por ejemplo, con el peso de un plato de un disco duro moderno de un equipo de cómputo personal de un terabyte (los discos duros donde se almacena la información están compuestos de uno o más platos circulares que rotan a gran velocidad mientras una o varias agujas se encargan de escribir o extraer datos de ellos). Un solo terabyte de capacidad podría pesar aproximadamente 50 gramos. Debido a que en 455 exabytes que es la capacidad de almacenamiento de un solo gramo de ADN, se pueden almacenar 455 millones de terabytes (Church, Gao, y Kosuri, 2012), y puesto que cada exabyte puede contener un millón de terabytes, La comparación inmediata es que en un solo gramo de ADN se puede almacenar el equivalente a 450 millones de discos duros, lo que es una cifra sorprendente. De cara a la durabilidad en el tiempo, positivas son las noticias de la posibilidad a futuro de hacer almacenamiento de información en moléculas de ADN, pues la información allí almacenada en condiciones adecuadas podría durar miles de años como ya se pudo determinar en el caso del genoma extraído de un caballo, que se comprobó estuvo en perfectas condiciones unos 700.000 años (Stefano, et al., 2018, p.2). De otro lado, están los procesos requeridos para el almacenamiento confiable, como por ejemplo la amplificación por PCR (de las siglas en inglés Polymerase Chain Reaction), técnica usada en biotecnología para clonar una molécula de ADN origen en muchos millones de moléculas ADN que son copias idénticas. Estas técnicas facilitarán el acceso posterior a la información al permitir su extracción para la lectura, pues una sola molécula de ADN es tan pequeña que en la práctica no es manejable. Pero a pesar de los grandes beneficios que estas técnicas moleculares prestarían a los procesos de almacenamiento de información sobre moléculas de ADN, su alto costo crea retos adicionales a mediano y largo plazo para sacar estas tecnologías de los laboratorios y así integrarlas en el manejo de datos de forma conveniente. Representación de datos y capacidades de almacenamiento ¿Cuánto puede ser comprimida la información usando ADN? Si cambiamos nuestra representación de datos de un sistema tradicional a un sistema basado en moléculas de ADN, la compresión va a depender de los modelos de representación. En un sistema binario, cada elemento mínimo de representación, o bit, es un 1 o un 0; esto exige mucho espacio de almacenamiento, ya que para cualquier símbolo o letra que se desee almacenar, debemos generar códigos de equivalencia. Por ejemplo, la letra ‘A’ en la tabla de equivalencias denominada ASCII es el código 65, y para almacenar dicho código necesitamos la secuencia binaria 8 bits 01000001. Así las cosas, asumiendo que la representación se haría usando 8 bits, tendríamos un máximo de 256 caracteres (códigos 0 a 255), que corresponden a las posibles combinaciones de ceros y unos entre 00000000 y 11111111. Hoy día existen otras representaciones más amplias (de tamaño variable), que permiten ampliar a muchos más bits la representación de distintos caracteres para obtener tablas de equivalencias mucho más amplias que la ASCII, en las que se podría representar cualquier carácter de cualquiera de los lenguajes escritos hoy conocidos. Dado que el ADN se compone de 4 nucleótidos (moléculas a partir de las cuales se construyen cadenas de ADN) Podría haber varias estrategias diferentes para representar datos en ella. La que primero se viene a la cabeza de muchos científicos y expertos en informática, es usar cada uno de los 4 posibles nucleótidos como una posición de un código cuaternario, así como en el sistema binario un bit representaba dos posibles valores. Eso mejora la codificación de los datos, ya que en una posición no tendríamos 2 potenciales valores 0 o 1 como en el sistema binario, sino 4 posibles valores (A, C, G y T), los cuales podríamos hacer corresponder, por ejemplo, con los valores 0, 1, 2 y 3 respectivamente, y así aumentar la capacidad de representación en un menor espacio. Pero una representación directa no es tan simple. Existen gran cantidad de problemas para la representación de datos, aunque en la misma medida van apareciendo nuevos modelos de representación, como lo ilustran Church, Gao y Kosuri (2012):
En cualquier caso, las magnitudes de almacenamiento de información usando moléculas de ADN son sobresalientes, considerando que en el año 2012 ya se podían obtener densidades de 5.5 petabits por cada milímetro cúbico, lo que representa un avance importante para ADN “suitable for immutable, high-latency, sequential access applications such as archival storage” (Church, et al., 2012, pag.1). Aparte del problema de representación de la información, otro reto que debe ser abordado cuando se trabaja con DNA es la longitud de las hebras. Grandes cantidades de información requieren largas cadenas de ADN. Sin embargo, sintetizar largas cadenas de ADN establece inconveniencias para las tecnologías de síntesis, pues no es posible sintetizar hebras de ADN de gran tamaño. Este problema ha sido abordado sintetizando cadenas de ADN cortas in vitro. De hecho, el ADN bien sintetizado puede tener larga vida incluso en ambientes de bajo mantenimiento. Por otro lado, las técnicas in vitro presentan desventajas de confiabilidad, escalabilidad, y costo-beneficio debido a las restricciones sobre los elementos del genoma y las localizaciones que pueden ser manipuladas, y que no generen un problema de visibilidad (Goldman, 2013, p.1). Una comparación, entre los mecanismos tradicionales de almacenamiento como cintas o discos magnéticos frente al las ventajas de almacenamiento en cadenas de nucleótidos o ADN, la expone Goldman (2013):
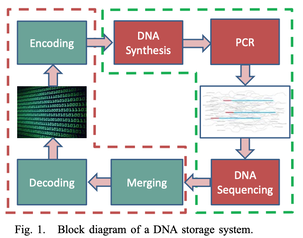
El impacto del crecimiento de los datos desde que apareció la computación en la nube es abrumador: alcanzó para el año 2018 media decena de zettabytes, unidades que no estamos acostumbrados a manejar. Para llevarse una idea, hoy los discos duros que pueden ser adquiridos en el Mercado rondan entre 1 y 4 terabytes, que es una gran capacidad. Un zettabyte equivale a mil millones de terabytes, y para ese año, 2018, ya existían aproximadamente 4.4 zettabytes cargados en la nube (Panda, 2018, p.1). Dados estos crecimientos, para dentro de un par de decenios la demanda de dispositivos de almacenamiento desbordará la capacidad de fabricarlos, y por eso es importante buscar otros medios de almacenamiento. Aunque las demandas de dispositivos con la tecnología actual pudieran ser hipotéticamente cubiertas, la durabilidad en el tiempo no está garantizada sino por algunos años. La molécula de ADN cubriría esta demanda, como nos planteaPanda (2018), al decir que el “DNA has a significantly higher longevity as a data storage molecule and can be easily amplified by polymerase chain reaction techniques to get the desired number of its copies”. Técnicas de representación e la información Un sistema diseñado para almacenamiento en DNA es descrito en líneas generales por Goela y Bolot (2016): La dificultad para sintetizar grandes secuencias de ADN a partir de nucleótidos individuales para construir una nueva molécula se debe abordar desde una representación de una cadena larga hipotética simulada directamente en computador. Los tipos de archivos de datos que se pueden tomar como base para ser representados en un esquema molecular de ADN, son de cualquier tipo entre los que tradicionalmente usamos cuando interactuamos con un computador personal, incluidos archivos de texto, PDFs, imágenes JPEG, y música en formato MP3. Los bytes que representan un archivo cualquiera en alguno de estos formatos se transforman a un sistema en base 3, en el que sólo se usan los dígitos 0, 1, 2, 3 (y no base 10 en el que se usan los dígitos del 0 al 9), donde cada dígito se hace equivaler a un nucleótido concreto (entre A, T, C, G), y que son la materia prima para hacer ADN. El texto completo se representa en una secuencia ADN simuladas en computador, y allí se simula su fragmentación, de tal forma que se solapan fragmentos consecutivos con redundancia de información, y también se adicionan datos en los extremos de los fragmentos moleculares simulados, que ayudan a la identificación de su posición en el archivo original (índices). Sigue llevar las moléculas desde un modelo computacional, a una molécula real de ADN. Cada fragmento es sintetizado usando biotecnología de síntesis de ADN. Las cadenas se deshidratan y se almacenan a temperatura ambiente, y para extraer la información codificada, se hace la codificación del texto desde la lectura del material genético, y el material genético se vuelve a almacenar (Goldman et al., 2013). Este modelo, que es el que se plantea en Goldman et al. (2013), especifica que la recuperación de la información se hace de forma técnicamente secuencial. Esto es, los índices solo sirven para ordenar la información. Sin embargo, los modelos actuales de acceso van mucho más allá, y requieren de técnicas más sofisticadas para poder llegar rápido a ella. En la investigación llevada a cabo por Bornholt et al. (2016), se planeta un modelo donde se usa un repositorio de ADN que hace un mapa de palabras clave sobre un volumen de información de ADN. En él, la unidad básica de almacenamiento es una cadena de ADN de entre 100 y 200 nucleótidos de largo, capaces de almacenar entre 50 y 100 bits en total. Allí se usan pares de valores, en los que un valor se usa como clave de búsqueda, y un segundo valor es una posición que indica dónde en el repositorio de cadenas de ADN se encuentra la información buscada. Este modelo es un mecanismo capaz de recuperar porciones de ADN de forma selectiva desde un repositorio contenedor de ADN con la información. Se parece a un índice alfabético de un libro, y, en este sentido, el modelo de Bornholt et al. (2016) se aproxima un poco más a la forma en que trabaja un dispositivo de almacenamiento tradicional actual. En lo pertinente a la representación de la información, el esquema de Bornholt y sus colaboradores es similar al planteado por Goldman y su equipo, en el que se aplica un sistema cuaternario de 4 dígitos 0, 1, 2, 3, siendo los dígitos los nucleótidos A, C, G y T. Sin embargo, dado que en la síntesis y en el secuenciado de moléculas de ADN puede haber errores, el modelo de Bornholt plantea una modificación adicional en la que se usa un modelo no cuaternario, sino terciario de sólo 3 dígitos, para dejar un cuarto dígito para un esquema de manejo de errores (Bornholt et al., 2016). Conclusión La era digital trajo consigo una dinámica sin precedentes en las comunicaciones y en la representación de datos, que llevó a un crecimiento exponencial en las necesidades de almacenamiento de la información. Sin embargo, los avances tecnológicos en los medios de almacenamiento electrónico actuales no son lo suficientemente robustos en términos de capacidad, velocidad y durabilidad en el tiempo, como para soportar el crecimiento explosivo de datos. Es imperativo considerar nuevas tecnologías y nuevos materiales que puedan suplir dichas deficiencias: una alternativa que se viene revisando, es el uso de moléculas de ADN como medio de almacenamiento masivo, pues ha mostrado poseer algunas de las características requeridas, en lo particular una alta capacidad de almacenamiento, y una alta resiliencia a distintos ambientes. La molécula de ADN se plantea entonces como un medio potencial para almacenar datos que no tengan que ser modificados con regularidad, o que sólo necesiten ser leídos, y actualmente hay muchos estudios experimentales alrededor del tema del uso de esta molécula como medio para almacenar y preservar información durante cientos o miles de años. Ha un tema concreto que se sale actualmente del radar del uso del ADN como medio, y es la velocidad de acceso, característica que podría llegar a ser cubierta por la biotecnología del futuro, de tal suerte que se habiliten altas velocidades de acceso y modificación a moléculas de ADN. Es altamente probable entonces que, por el momento, se empiecen a dar tecnologías híbridas entre las tecnologías actuales, y el uso de nuevos materiales que podrían descubrirse, donde una de esas alternativas es la molécula de la vida, el ADN. Fuentes Bornholt, J., Lopez, R., Carmean, D. M., Ceze, L., Seelig, G., & Strauss, K. (2016). A DNA-Based Archival Storage System. Proceedings of the Twenty-First International Conference on Architectural Support for Programming Languages and Operating Systems - ASPLOS ’16, 637–649. https://doi.org/10.1145/2872362.2872397 Church, G. M., Gao, Y., & Kosuri, S. (2012). Next-Generation Digital Information Storage in DNA. Science, 337(6102), 1628–1628. https://doi.org/10.1126/science.1226355 Dodd, M. S., Papineau, D., Grenne, T., Slack, J. F., Rittner, M., Pirajno, F., O’Neil, J., & Little, C. T. S. (2017). Evidence for early life in Earth’s oldest hydrothermal vent precipitates. Nature, 543(7643), 60–64. https://doi.org/10.1038/nature21377 International Human Genome Sequencing Consortium. (2001). Initial sequencing and analysis of the human genome. Nature, 409(6822), 860–921. https://doi.org/10.1038/35057062 Goldman, N., Bertone, P., Chen, S., Dessimoz, C., LeProust, E. M., Sipos, B., & Birney, E. (2013). Towards practical, high-capacity, low-maintenance information storage in synthesized DNA. Nature, 494(7435), 77–80.https://doi.org/10.1038/nature11875 Goela, N., & Bolot, J. (2016). Encoding movies and data in DNA storage. 2016 Information Theory and Applications Workshop (ITA), 1–1. https://doi.org/10.1109/ITA.2016.7888163 Mandelkern, M., Elias, J. G., Eden, D., & Crothers, D. M. (1981). The dimensions of DNA in solution. Journal of Molecular Biology, 152(1), 153–161. https://doi.org/10.1016/0022-2836(81)90099-1 Panda, D., Molla, K. A., Baig, M. J., Swain, A., Behera, D., & Dash, M. (2018). DNA as a digital information storage device: Hope or hype? 3 Biotech, 8(5), 239. https://doi.org/10.1007/s13205-018-1246-7 Stefano, G. B., Wang, F., & Kream, R. M. (2018). DNA MemoChip: Long-Term and High Capacity Information Storage and Select Retrieval. Medical Science Monitor, 24, 1185–1187. https://doi.org/10.12659/MSM.908313 Watson, J. D., & Crick, F. H. C. (1953). Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. Nature, 171(4356), 737–738. https://doi.org/10.1038/171737a0
1 Comment
Leave a Reply. |
Categories
All
Archives
July 2020
|

 RSS Feed
RSS Feed